制限酵素
ミトコンドリア・イブの話しでは、mtDNAの解析は12の「制限酵素」で切断した断片の有無が比較されたらしい。パズル的な関心で調べてみる。
これはWikipediaによれば、II型制限酵素の話しであるらしい。
Fig.1 12の制限酵素
| 制限酵素 |
| インフルエンザ菌 |
GTTAAC |
---GTT AAC--- |
| 腸炎ビブリオ |
GGACC
GGTCC |
---G GACC---
---G GTCC--- |
バチルス・ステアロサーモフィルス
(好熱性細菌) |
CGCG |
|
| ヘモフィラス・ヘモリティカス |
GCGC |
|
| インフルエンザ菌 |
CCGG |
---C CGG--- |
| モラクセラ・vobis |
GATC |
--- GATC--- |
| 好熱菌 |
TCGA |
---T CGA--- |
| ロドシュードモナス・スフェロイデス |
GTAC |
---GT AC--- |
| インフルエンザ菌 |
GAATC
GAGTC
GACTC
GATTC |
---G AATC---
---G AGTC---
---G ACTC---
---G ATTC--- |
| ヘモフィルス・egytius |
GGCC |
---GG CC--- |
| アースロバクタ・ルテウス |
AGCT |
---AG CT--- |
デスルホビブリオ・デスルフリカンス
(硫酸還元菌の一種) |
CTAAG
CTGAG
CTCAG
CTTAG |
---C TAAG---
---C TGAG---
---C TCAG---
---C TTAG--- |
-
5'→3' で記す。
-
--- は、塩基配列が続いていることを示す。
回文(Palindrome)
-
5’端、3’端
ヌクレオチドは、塩基-糖-リン酸 の構造をしており、リン酸によって結合する。
リン酸は、糖(5炭糖:ペントース)の5位の炭素のOH基の位置に付いている。
このリン酸が別のヌクレオチドの糖の3位の炭素のOH基の位置に結合して鎖状になる。
この方向性を、5'---AGCT---3' のように表記することがある。
通常は、5'→3' の向きに書かれる。
-
塩基配列の端
始点(5'末端)は、糖にOH基があるリン酸が付いている。
終点(3’末端)の糖の3'には、リン酸ではなく、OH基が付いている。
-
mtDNAの末端
環状鎖の第1座位はどのように決められているか。
わからない。3’末端などが使われているが、分子構造上は閉環で、末端がないもだと思う。座位16,569のヌクレオチドは、座位1のヌクレオチドとつながっている。
-
H鎖、L鎖
制限酵素は、2本鎖を切断する。mtDNAは、H鎖とL鎖からなる。
-
相補鎖
H鎖とL鎖は相補鎖を成している。L鎖の塩基配列は、H鎖の塩基配列に対して、AとT、GとCの相補塩基対の関係にある。
5'GTTAAC は、3'CAATTG とが対になっている。
・5’端、3’端のについては、H鎖とL鎖は逆向きに結合している。
・制限酵素は相補鎖を切るので認識位置は5'側からの1通りを示せば良いようだ。
・GTTAAC の相補鎖を問われたら、GTTAAC と答えるのが常識らしい。
- 5'GTTAAC と3'CAATTG とが対になるので、パリンドローム(回文)と言うらしい。
この関係は、「多くは」が付いているので、制限酵素の認識配列全てではないらしい。
どうやら、制限酵素は回文になっている場所を認識して切断すると言うことで、相補鎖全体が回文になっているわけでははい。最初の塩基と最後の塩基には関連がないので当然だ。
では、どんな場所が回文になるのか。
これは、偶数の組でしか起きないのはわかる。必ず塩基対の記号は異なるので、奇数では回文にならない。HinfI の場合、GAnTC を認識するが回文にはならない。
これ以上は考えられないので書き出してみる。短いものは長いものの部分であり得、重複してカウントされている。最大は16文字だった。(7328 COX1)
fig.2 rCRS mtDNAの回文パターン
| 回 |
| cg |
1 |
acatgt |
3 |
gtatac |
1 |
aaaatttt |
1 |
taatatta |
1 |
aagacgtctt |
| gc |
1 |
agcgct |
3 |
gttaac |
1 |
aactagtt |
1 |
tattaata |
1 |
aagtatactt |
| at |
1 |
atcgat |
3 |
tccgga |
1 |
aaggcctt |
1 |
tcaattga |
1 |
aagtgcactt |
| ta |
1 |
cagctg |
4 |
ccatgg |
1 |
accgcggt |
1 |
tcatatga |
1 |
aataattatt |
| cgcg |
1 |
cggccg |
4 |
tgatca |
1 |
actatagt |
1 |
tgagctca |
1 |
atgatatcat |
| gcgc |
1 |
cgtacg |
4 |
ttgcaa |
1 |
agaattct |
2 |
ataattat |
1 |
attaattaat |
| acgt |
1 |
ctcgag |
4 |
tttaaa |
1 |
agacgtct |
2 |
atagctat |
1 |
cagggccctg |
| ccgg |
1 |
cttaag |
5 |
agtact |
1 |
aggatcct |
2 |
ctcatgag |
1 |
cctctagagg |
| gatc |
1 |
gcatgc |
5 |
atatat |
1 |
agggccct |
2 |
ctctagag |
1 |
gaattaattc |
| tcga |
1 |
gccggc |
5 |
caattg |
1 |
agtatact |
2 |
cttcgaag |
1 |
taattaatta |
| gtac |
1 |
gctagc |
5 |
gggccc |
1 |
agtgcact |
2 |
tactagta |
1 |
tattataata |
| tgca |
1 |
ggatcc |
5 |
tcatga |
1 |
atggccat |
2 |
tcctagga |
1 |
tgaagcttca |
| catg |
1 |
gtgcac |
5 |
tctaga |
1 |
attataat |
2 |
tgatatca |
1 |
ttcctaggaa |
| ggcc |
1 |
tacgta |
5 |
tggcca |
1 |
caacgttg |
3 |
aattaatt |
2 |
atactagtat |
| agct |
2 |
aacgtt |
5 |
ttataa |
1 |
caatattg |
4 |
taggccta |
2 |
gcttcgaagc |
| atat |
2 |
ccgcgg |
6 |
tagcta |
1 |
cagtactg |
4 |
ttaattaa |
1 |
caagacgtcttg |
| tata |
2 |
ctgcag |
7 |
ctatag |
1 |
ccaattgg |
|
|
1 |
cgcttcgaagcg |
| aatt |
2 |
gacgtc |
7 |
tatata |
1 |
ccctaggg |
|
|
1 |
ggcttcgaagcc |
| ctag |
2 |
gagctc |
7 |
ttcgaa |
1 |
cgatatcg |
|
|
1 |
taataattatta |
| ttaa |
2 |
ggcgcc |
8 |
aaattt |
1 |
ctgatcag |
|
|
1 |
ttaattaattaa |
|
3 |
aagctt |
9 |
actagt |
1 |
gaagcttc |
|
|
1 |
ttattataataa |
|
3 |
atgcat |
10 |
attaat |
1 |
gccatggc |
|
|
1 |
tcgcttcgaagcga |
|
3 |
catatg |
11 |
aatatt |
1 |
gctatagc |
|
|
1 |
ttcgcttcgaagcgaa |
|
3 |
gaattc |
12 |
aggcct |
1 |
gtaattac |
|
|
|
|
|
3 |
gatatc |
12 |
cctagg |
1 |
gtttaaac |
|
|
|
|
|
3 |
ggtacc |
14 |
taatta |
1 |
taaattta |
|
|
|
|
- string ComplementaryPairString()
- {
- char[] p = new char[HomoSapiensMitochondrion.Origin.Length];
- for (int i = 0; i < p.Length; i++)
- {
- char c = HomoSapiensMitochondrion.Origin[i];
- switch (c)
- {
- case 'a': p[i] = 't'; break;
- case 't': p[i] = 'a'; break;
- case 'c': p[i] = 'g'; break;
- case 'g': p[i] = 'c'; break;
- default: p[i] = c; break;
- }
- }
- return new string(p);
- }
- void palindromic()
- {
- // 環状なのでそれそれ2つを連結して終端部分を特別に処理しなくても良いように
- string H鎖 = HomoSapiensMitochondrion.Origin;
- H鎖 = H鎖.Replace("n", ""); // 3107の欠番除去。0-16567で使う。
- int 鎖長 = H鎖.Length; // 16568
- H鎖 += H鎖;
- string L鎖 = ComplementaryPairString();
- L鎖 = L鎖.Replace("n", ""); //3107の欠番除去。0-16567で使う。
- L鎖 += L鎖;
- // 一致箇所の記録
- SortedList<string, int> 記録 = new SortedList<string, int>();
- // 1塩基ずつ進める
- int p = 鎖長;
- for (int i = 0; i < 鎖長; i++)
- {
- // 最遠端から回文になっていないか調べる
- for (int j = 0; j < 鎖長; j++)
- {
- bool isPalindrome = true;
- for (int k = 0; k < 鎖長 - j; k++)
- {
- if (H鎖[i + k] != L鎖[i + 鎖長 - j - k - 1])
- {
- isPalindrome = false;
- break;
- }
- }
- if (isPalindrome)
- {
- // 回文になっている
- int 一致した長さ=鎖長 - j;
- string key = H鎖.Substring(i, 一致した長さ) + " "
- + L鎖.Substring(i, 一致した長さ);
- if (記録.ContainsKey(key))
- {
- 記録[key]++;//記登録。カウントアップ
- }
- else
- {
- 記録.Add(key, 1);//未登録。追加する。
- }
- }
- }
- }
- foreach(string key in 記録.Keys)
- {
- Debug.WriteLine(記録[key] + " " + key);
- }
- }
どんな箇所でどれぐらいに切れるのか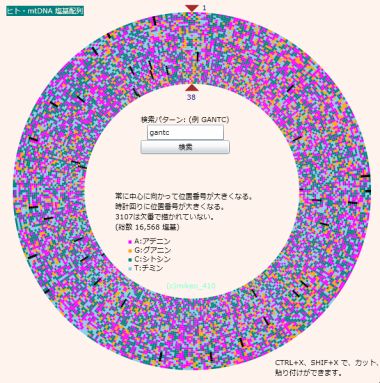
Fig.3 制限酵素の認識箇所の数
| 制限酵素 |
| GTTAAC |
3 |
GGACC
GGTCC |
8 |
| CGCG |
6 |
| GCGC |
17 |
| CCGG |
23 |
| GATC |
23 |
| TCGA |
29 |
| GTAC |
35 |
| GAnTC |
36 |
| GGCC |
49 |
| AGCT |
64 |
| CTnAG |
72 |
| 計 |
365 |
「gantc」を検索してみた。左図の黒い印が認識サイト。
すべて試すと計365となった。
ミトコンドリア・イブの話しでは、この12の制限酵素で、147人のサンプルから467サイトが確認された。平均370サイト、195の多様性があった。195について、少なくとも1人は持っていない人がいることを意味する。これをもとに 133 の型にわけられた。
このことから、各人については、272サイトほどは共通で、98サイトほどが他の人と一致しない可能性のある部分と言うことだと思う。
制限酵素と塩基番号
Fig.4 制限酵素の認識と塩基番号
| 制限酵素 |
|
|
| 8249 |
W |
|
|
|
|
|
|
|
|
|
|
|
|
12308
16065 |
U,K
J |
|
|
7025
10028
10397
15606 |
H
I
M
T |
1715
10394 |
X
H,T,U,V,W,X |
制限酵素を使った分析からわかる突然変異箇所の塩基番号とハプログループに関する記述を拾っておく。
AvaII を見てみる。rCRSで検索すると、認識位置は以下のようになる。
塩基番号8249とは無縁なようだ。
ggacc 3 箇所の一致
1169 ggacc
2268 ggacc
2621 ggacc
ggtcc 5 箇所の一致
657 ggtcc
2776 ggtcc
12629 ggtcc
13367 ggtcc
16390 ggtcc | ハプログループ W を見ると、G8251Aがある。
どうやら、G->Aの変異があると、8249が認識サイトになると言うことのようだ。
8249は、遺伝子 COX2 にある。アミノ酸をコードしている。
Fig4.1 のように 8249 はトリプレットの先頭で、実際の変異箇所が 8251 と言うことらしい。
この変異は、1塩基置換で、変異前(ggg)も変異後(gga)も G(グリシン) を指す同義置換。
Fig.4.1 G8251C の前後
| 塩基番号 |
| a |
|
M |
| t |
|
| a |
|
| g |
|
G |
| g |
|
| g |
a |
| c |
|
P |
| c |
|
| c |
|
変異の結果、8249 から、ggacc となるので、AvaII の認識サイトになる。
おそらく遺伝子上の変異なので早くから知られ、アミノ酸の位置で呼ばれていたものに、塩基番号を対応させて 8249 と記していたものと推測する。
rCRSなら 8 に分割されるものが、この変異で9に分割されることになり、rCRSと一致するものが7、一致しないものが2できると言ったことが想像できる。
やはり、制限酵素の話しは、全塩基配列を比較する話しとは同じでないように思える。
各部の名称と塩基配列
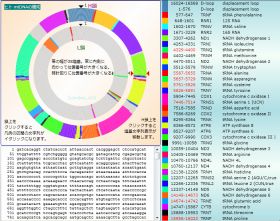 mtDNAは、閉環状2重鎖で、H鎖(重鎖)、L鎖(軽鎖)からなる。 mtDNAは、閉環状2重鎖で、H鎖(重鎖)、L鎖(軽鎖)からなる。
NBCIのデータには、部位の名前らしいものが付いている。それ以外にも呼び名があるようだ。
2つのリボソームRNA、22のtRNA、13のタンパク質を指す37の遺伝子がある。
それぞれの部位は、648..1601 RNR1 のように、塩基番号で示されている。
相補鎖
| 塩基番号 |
| 5' |
g |
a |
t |
- |
a |
g |
t |
c |
- |
g |
3' |
| 3' |
a |
g |
c |
- |
g |
a |
c |
t |
- |
a |
5' |
H鎖の相補鎖がL鎖で、閉環状を成している。ヌクレオチドの接続状態には特別な差異はないようで、どうやって塩基番号1を決めているのかはわからない。
5'端、3'端の分子の向きが異なる。このことは、トリプレットを解釈する際に意味を持つ。H鎖、L鎖とも同じコード表を使って変換するなら、H鎖上の gat の箇所は、agc ではなく cga を引く必要がある。
-
これは、文字列を処理する話しで、化学的にどうなっているかはわからない。
おそらく、どの鎖にあるかにかかわらず、同じ反応が起きているだけなのだと推測する。
-
一方の鎖上で有効なトリプレットの相補鎖側も有効なトリプレットであるかはわからない。
タンパク質の合成に作用するのは、一方の鎖のみであるようだ。
-
遺伝子領域を見つけると言った場合は、L鎖の配列の並びを反転させれば、H鎖と同じ処理ができる。
ただし、塩基番号の表記は維持しなければならない。
-
ND6 の部分は、 complement(14149..14673) と記されている。
L鎖上を、14673 -> 14149 に処理すれば、アミノ酸配列が得られることがわかる。
突然変異の表記
塩基番号は1から16,569で表され、3107は欠番(塩基番号は存在するが塩基は割り当てない)。
ただし、実際のサンプルを調べれば、長さが異なり、どこが3107なのかも直ちにはわからない。
塩基番号は、rCRS の塩基配列に基づいている。差異は数パーセント以下なので、rCRSを基準にして、3107の位置や挿入欠失も表せる。
ただし、ハプロタイプに関連した話題の場合で、医療など変異自体の性質の話しでは、rCRS と比較しなければならない必然性はない。
A750G は、塩基番号750のAがGに変わる変異を表すようだ。塩基番号309.1Cは、309にCが挿入される変異を表している。
この表記は、考えてみると難しい。
-
750 が G のサンプルがあったとして、元がなんであるかがわからない。
-
2つの塩基を比較して、挿入欠失がわかったとして、どちらの挿入か欠失かはわからない。
rCRSが基準なら
読み方に注意が必要だ。
-
差異は必ずしもサンプルの突然変異を表さない。
検出される315.1C は、rCRSの欠失による。サンプルに挿入があったことを示さない。
-
A263Gは多くのサンプルで差異として検出されるが、この変異はH2a2 を特徴づけるもので、H2aに起きた変異で、サンプルの突然変異ではない。
-
A750G の Aは、1)rCRS のAなのか、2)サンプルのハプロタイプに基づく推定なのか。
-
サンプルにある突然変異を選別するには、変異の生起順や系統がわかっている必要がある。
突然変異の確率や遺伝子距離などは確証を与えない。
健常者が基準なら
何が健常者かはともかく、医療では集団内の分布状況が注目されるものと想像する。「孤発性患者の50%以上でxxx遺伝子に突然変異が認められる。」のような場合の「突然変異」は、大多数の人と異なると言うことだと思う。
こうした話しでは、遺伝子名や並列を表記する方法が採られ、必ずしも塩基番号を使う必要性はない。
実際、750Gのような記法は使われないようだ。
ハプロタイプを決めるなら
ハプロタイプを決めるためなら、特徴となる塩基番号と塩基を記せば良い。
この場合は、前の塩基は出てこないので、263G のような記述になる。実際には、番号だけのものもある。
これは、検査方法が何かとの比較を行うもので、差異として検出された場所だけが判定に必要だからだと推測する。
rCRSの突然変異
rCRSは、ハプログループ H2a2 であるらしい。これと比較する場合は、H2a2の変異を除いてみる必要がある。
Fig.5 rCRS の遺伝子と変異(L3以降)
| 位置 |
| 16569 |
D-loop |
HVR1 |
調節領域 |
16223T(H) |
| 576 |
HVR2 |
73A(R)
263A(H2a2)
309.1C(?)
315.1C(H) |
| 647 |
TRNF |
|
tRNA phenylalanine |
|
| 1601 |
RNR1 |
12S |
リボソームRNA |
750A(H2a)
1438A(H) |
| 1670 |
TRNV |
|
tRNA valine |
|
| 3229 |
RNR2 |
16S |
リボソームRNA |
2706A(HV)
3010G(H2a2)
3107(非存在)
|
| 3304 |
TRNL1 |
|
tRNA leucine 1 (UUA/G) |
|
| 4262 |
ND1 |
|
NADHデヒドロゲナーゼ 1 |
|
| 4331 |
TRNI |
|
tRNA isoleucine |
|
| 4400 |
TRNQ |
|
tRNA glutamine |
|
| 4469 |
TRNM |
|
tRNA methionine |
|
| 5511 |
ND2 |
|
NADHデヒドロゲナーゼ 2 |
4769A(H2) |
| 5579 |
TRNW |
|
tRNA tryptophan |
|
| 5655 |
TRNA |
|
tRNA alanine |
|
| 5729 |
TRNN |
|
tRNA asparagine |
|
| 5826 |
TRNC |
|
tRNA cysteine |
|
| 5891 |
TRNY |
|
tRNA tyrosine |
|
| 7445 |
COX1 |
|
cytochrome c oxidase I |
7028C(HV) |
| 7514 |
TRNS1 |
|
tRNA serine 1 (UCN) |
|
| 7585 |
TRND |
|
tRNA aspartic acid |
|
| 8269 |
COX2 |
|
cytochrome c oxidase II |
|
| 8364 |
TRNK |
|
tRNA lysine |
|
| 8572 |
ATP8 |
|
ATP synthase 8 |
|
| 9207 |
ATP6 |
|
ATP synthase 6 |
8860A(H2a2) |
| 9990 |
COX3 |
|
cytochrome c oxidase III |
|
| 10058 |
TRNG |
|
tRNA glycine |
|
| 10404 |
ND3 |
|
NADHデヒドロゲナーゼ 3 |
|
| 10469 |
TRNR |
|
tRNA arginine |
|
| 10766 |
ND4L |
|
NADHデヒドロゲナーゼ 4L |
|
| 12137 |
ND4 |
|
NADHデヒドロゲナーゼ 4 |
11719G(R) |
| 12206 |
TRNH |
|
tRNA histidine |
|
| 12265 |
TRNS2 |
|
tRNA serine 2 (AGU/C) |
|
| 12336 |
TRNL2 |
|
tRNA leucine 2 (CUN) |
|
| 14148 |
ND5 |
|
NADHデヒドロゲナーゼ 5 |
12705C(R) |
| 14673 |
ND6 |
|
NADHデヒドロゲナーゼ 6 |
|
| 14742 |
TRNE |
|
tRNA glutamic acid |
|
| 15887 |
CYTB |
|
cytochrome b |
14766C(R0)
15301G(N)
15326A(H2a2) |
| 15953 |
TRNT |
|
tRNA threonine |
|
| 16023 |
TRNP |
|
tRNA proline |
| |